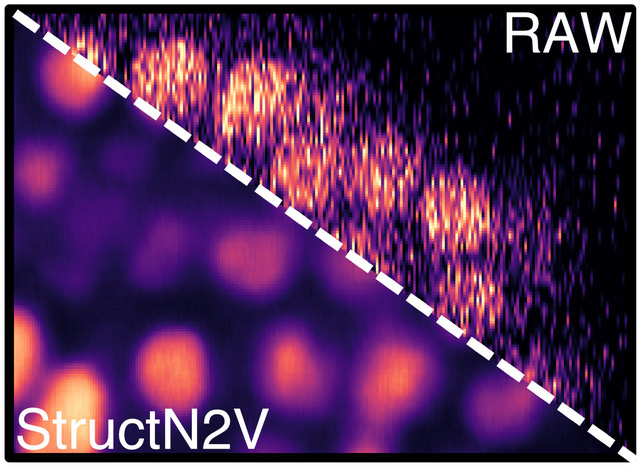
Die selbstkontrollierte StructN2V entfernt das vertikale Streifenrauschen aus dem konfokalen Bild des sich entwickelnden C. elegans-Embryos. © Waterston Lab, University of Washington, Seattle, WA, USA via celltrackingchallenge.net
Die Fluoreszenzmikroskopie ist zu einem unverzichtbaren Werkzeug für die Beantwortung grundlegender Fragen in den biomedizinischen Wissenschaften geworden. Solche Mikroskope visualisieren die Position von fluoreszenzmarkierten Zellen in biologischen Geweben und können jede einzelne Zelle in ganzen, sich entwickelnden Organismen wie Würmern, Fischen oder Mäusen aufnehmen. Wie die normale Fotografie benötigt auch die Fluoreszenzmikroskopie eine ausreichende Lichtmenge. Die benötigte Lichtmenge kann jedoch leicht Werte erreichen, die unseren Modellorganismen schaden. Um dies zu vermeiden, versuchen wir, so wenig Licht wie möglich zu verwenden, was - genau wie bei der Nachtfotografie - Rauschen und eine körnige Textur in unseren Bildern verstärkt. Das ist ein großes Problem für Biologen, die versuchen zu erkennen und zu interpretieren, was die Zellen tun.
Die Fortschritte bei der Verwendung künstlicher neuronaler Netzwerke in den letzten Jahren haben zu einer Explosion von Bildwiederherstellungsmethoden zur Beseitigung von Rauschen und zur Verbesserung der Bildqualität geführt. Solche trainierten neuronalen Netzwerke können den Inhalt "enthüllen", der in Mikroskopie-Bildern von geringer Qualität verborgen ist. Sie können hochwertige Mikroskopie-Bilder erstellen, selbst wenn sie mit wenig Licht aufgenommen wurden.
Forscher der Gruppe um Gene Myers am MPI-CBG und des benachbarten Zentrums für Systembiologie Dresden (CSBD) haben nun den vielversprechenden "blinden Fleck"-Ansatz ihrer Kollegen aus der Gruppe von Florian Jug (CSBD und MPI-CBG) weiterentwickelt. Die neue Structured Noise2Void (STRUCTN2V)-Methode des Myers Teams erweitert den "blinden Fleck"-Ansatz, um eine größere Palette von Rauschen und unerwünschter Textur zu entfernen.
Coleman Broaddus, Erstautor der Studie, die kürzlich im Konferenzband des IEEE International Symposium on Biomedical Imaging (ISBI) veröffentlicht wurde, erklärt: "Ein Blinder Fleck-Netzwerk wird trainiert, indem man ihm ein Stück seines Bildes zeigt, aber ein Pixel in der Mitte (den blinden Fleck) versteckt und das Netzwerk bittet, seinen Wert zu erraten. Überraschenderweise haben wir festgestellt, dass das Rauschen, wenn es eine bestimmte Struktur hat, zusätzliche Pixel um den blinden Fleck herum sorgfältig verbirgt (wodurch dem Netzwerk noch weniger Informationen zur Verfügung stehen). Das führt dazu, dass es den richtigen Wert besser erraten kann."
Gene Myers, Direktor am CSBD und am MPI-CBG und Leiter der Studie, fügt hinzu: "Unser Ansatz passt gut zu den bestehenden Entrauschungsmethoden, die bereits in der Open-Source-Bibliothek "Noise2Void" (https://github.com/juglab/n2v) verfügbar sind, die vom Jug-Labor unterhalten und von Forschern auf der ganzen Welt genutzt wird. Wir möchten, dass die Leute unsere Methode mit so wenig Aufwand wie möglich an ihren Daten ausprobieren. Wir hoffen, dass wir durch die weite Verbreitung die Grenzen dieser Technik erforschen können, während wir gleichzeitig möglichst vielen Menschen helfen, die Qualität ihrer Mikroskopie-Bilder zu verbessern."
Coleman Broaddus, Alexander Krull, Martin Weigert, Uwe Schmidt, Gene Myers: „Removing Structured Noise With Self-Supervised Blind-Spot Networks“, Proceedings of the IEEE 17th International Symposium on Biomedical Imaging (ISBI). 2020. DOI: 10.1109/ISBI45749.2020.9098336
Link to open source library: https://github.com/mpicbg-csbd/structured_N2V_paper/