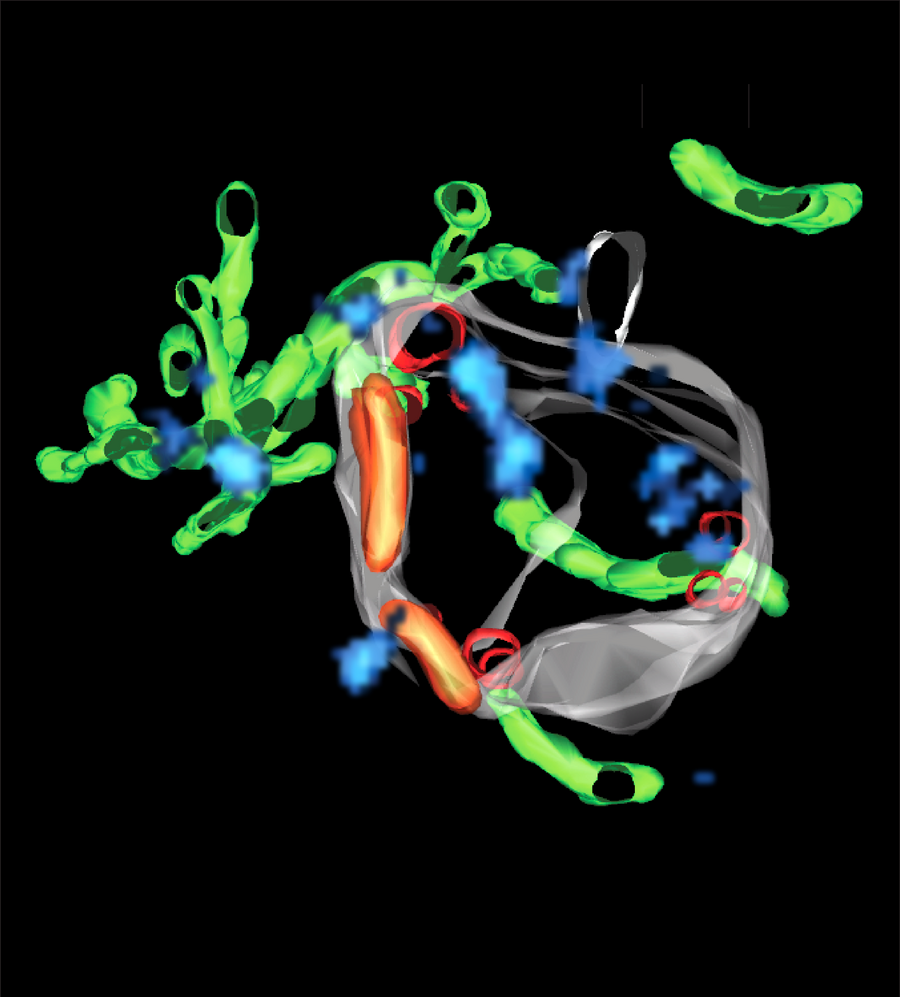
Wie man mit superCLEM ein Endosom sichtbar machen kann: Ein dreifarbiges SMLM-Bild und die entsprechende dreidimensionale Rekonstruktion des gesamten Endosoms, basierend auf einem Elektronentomogramm, werden verwendet, um ein detailliertes Modell eines frühen Endosoms im Nanobereich zu erstellen. Es zeigt verschiedene, strukturell unterschiedliche endosomale Nanodomänen, wie zum Beispiel kleine Vesikel im Inneren (rot) und Sortierbereiche an der Außenseite der endosomalen Membran (orange), feine Tubuli am endosomalen Körper (grün) und Rab5-Nanodomänen an der Oberfläche des Endosoms (blau).
Die Zelle birgt noch viele Geheimnisse in sich. Die Pionierarbeit der Elektronenmikroskopie in den 1960er Jahren machte Erkenntnisse zur Struktur und Funktion subzellulärer Organellen möglich. Seitdem haben Wissenschaftler große Fortschritte auf diesem Gebiet gemacht. Sie können nun erklären welche molekularen Mechanismen eine Rolle bei der Bildung solcher Organelle spielen und wie diese miteinander ‚sprechen‘. Zelluläre Organellen sind nicht nur ‚Säcke‘ mit Molekülen, sondern haben auch unterschiedliche Formen. Endosomen sind beispielsweise membrangebundene Organelle, deren Form einem Tintenfisch ähnelt, wobei die Tentakel Röhren repräsentieren, die dabei helfen Moleküle zurück an die Zelloberfläche zu bringen. Darüber hinaus funktionieren Endosomen der Oberfläche der Zelle, wo sie Moleküle in Empfang nehmen und sie innerhalb der Zelle verteilen, ähnlich wie auf einem Postamt.
Bisher war es nicht möglich, eine genaue Landkarte von Frachtmolekülen und solchen, die den Endosomen spezifische Funktionen zuweisen, zu erstellen. Dazu sind neue Methoden notwendig, mit denen man verschiedene Moleküle gleichzeitig visualisieren kann. Es gibt bereits mehrere leistungsstarke Methoden. In der Zellbiologie kommt die Lichtmikroskopie oft zum Einsatz, ist aber aufgrund der Lichtbeugung auf 200 Nanometer (0,0002 mm) Auflösung begrenzt. Daher können die Details vieler Organellen nicht gut aufgelöst und dargestellt werden. Ein weiteres Verfahren, die Einzelmolekül-Lokalisationsmikroskopie (SMLM), ist ein hochauflösendes bildgebendes Verfahren, das eine nahezu molekulare Auflösung bis zu 20 Nanometern (0,00002 mm) ermöglicht. Sie kann jedoch nicht die gesamte Struktur eines Organelles erfassen. Mit der Elektronenmikroskopie (EM) hingegen kann man die gesamte Struktur eines Organelles sehen, aber es ist schwierig, ihre molekularen Bestandteile zu veranschaulichen.
Um dieses Problem zu lösen, bildete Marino Zerial, Direktor am MPI-CBG, ein Team aus Zellbiologen und Licht- und Elektronenmikroskopikern. Das Team kombinierte Dreifarben-SMLM und dreidimensionale Elektronentomografie in einem neuartigen Ansatz, genannt superCLEM. Um die superCLEM-Methode zu demonstrieren, untersuchten die Forscher Endosomen und visualisierten mehrere Moleküle gleichzeitig, ohne die feine Struktur dieser Organellen zu zerstören. Christian Franke, Sandra Segeletz und Urska Repnik, drei der Autoren der Studie, erklären: „Bisher war es nicht möglich, mehrere bestimmte Proteine mit einer Auflösung im Nanometerbereich und im Kontext zueinander auf der dreidimensionalen Struktur eines Organelles gleichzeitig zu zeigen.“ Sie fahren fort: „Die wichtigste Erkenntnis der Studie ist, dass das Rab5-Protein, eine Hauptkomponente der Endosomen, nicht zufällig verteilt ist, sondern in kleinen regulatorischen Regionen konzentriert ist, die wie Kontrollzentren auf der Endosomen-Membran agieren. Das wurde so präzise noch nie zuvor gezeigt.“
Die superCLEM Methode eröffnet ein neues Feld, um Struktur-Funktionsanalysen an unterschiedlichsten Zellorganellen im Nanometerbereich zu erstellen. Das Verfahren ist sehr anpassungsfähig für viele verschiedene Situationen. 3D-SMLM-Methoden und alternative EM-Ansätze werden superCLEM in Zukunft noch weiter verbessern.
Franke, C, Repnik, U, Segeletz, S, et al. Correlative single‐molecule localization microscopy and electron tomography reveals endosome nanoscale domains. Traffic. 2019; 20: 601– 617.