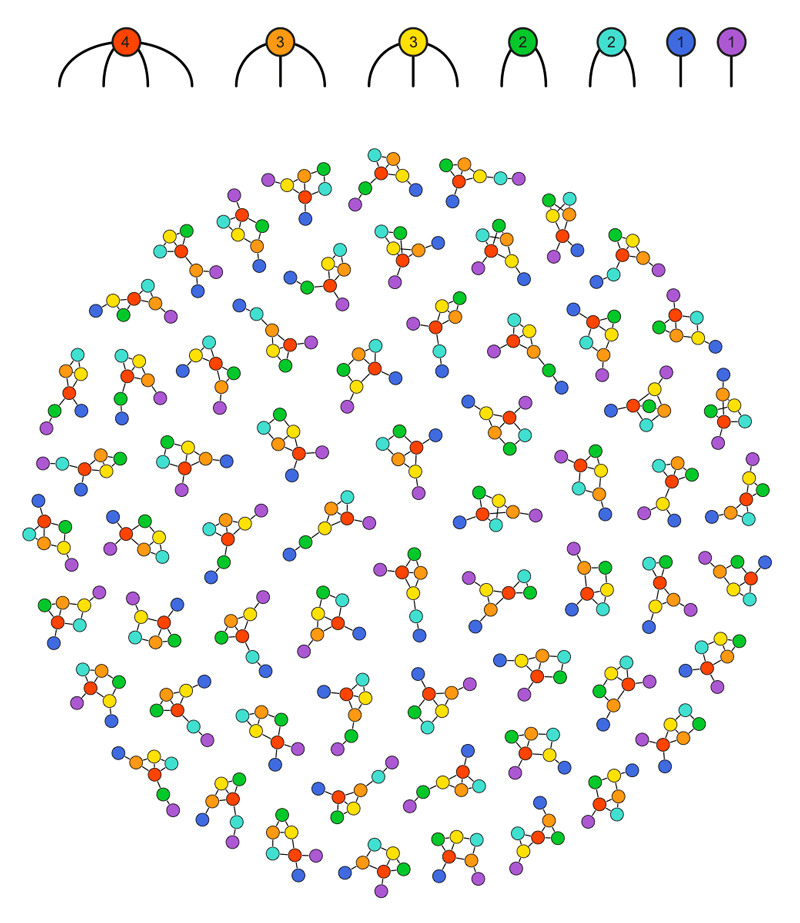
Die Farben stellen die Identitäten der Knoten dar. In der oberen Zeile sieht man, wie viele Verbindungen jeder Knoten hat. Im Kreis sind alle denkbaren Möglichkeiten dargestellt, aus diesen Knoten einen zusammenhängenden Graphen zu bilden. Der neue Algorithmus kann eine davon zufällig und wiederholt auswählen. © Szabolcs Horvat et al. 2020 / MPI-CBG / CSBD
Viele natürliche und von Menschen geschaffene Netzwerke, wie beispielsweise computerbasierte, biologische oder soziale Netzwerke, haben eine Vernetzung die ihr Verhalten entscheidend prägt. Das Fachgebiet der Netzwerkwissenschaft befasst sich mit der Analyse von solchen komplexen Netzwerken in der realen Welt und will verstehen, wie die Struktur von Netzwerken ihre Funktion oder ihr Verhalten beeinflusst. Beispiele sind das Gefäßnetzwerk unseres Körpers, das Netzwerk der Neuronen in unserem Gehirn oder die Ausbreitung einer Epidemie in einer Gesellschaft.
Die Notwendigkeit zuverlässiger Nullmodelle
Bei der Analyse derartiger Netzwerke geht es oft darum, interessante Eigenschaften und Merkmale zu finden. Trägt zum Beispiel die Struktur eines bestimmten Kontaktnetzwerks dazu bei, dass sich Krankheiten besonders schnell ausbreiten? Um das herauszufinden, braucht man eine Ausgangsbasis zum Vergleich – eine Menge von zufälligen Netzwerken, ein sogenanntes „Nullmodel.“ Da mehr Verbindungen deutlich mehr Möglichkeiten für eine Infektion schaffen, sollte die Anzahl der Verbindungen jedes Knotens im Vergleichsnetzwerk an das analysierte Netzwerk angepasst werden. Wenn dann dieses Netzwerk die Ausbreitung mehr zu erleichtern scheint als das Vergleichsnetzwerk, weiß man, dass es an seiner spezifischen Netzwerkstruktur liegen muss. Es ist allerdings schwierig, wirklich zufällige, unverfälschte Nullmodelle zu erstellen, die an bestimme Eigenschaften angepasst sind, und erfordert in der Regel einen anderen Ansatz für jede Eigenschaft von Interesse. Bestehende Algorithmen, die zusammenhängende Netzwerke mit einer bestimmten Anzahl von Verbindungen für jeden Knoten erstellen, leiden alle unter unkontrollierten Verzerrungen. Das bedeutet, dass einige Netzwerke öfter generiert werden als andere, was möglicherweise die Schlussfolgerungen der Studie beeinträchtigen könnte.
Eine neue Methode, die Verzerrungen beseitigt
Szabolcs Horvát und Carl Modes vom Zentrum für Systembiologie Dresden (CSBD) und dem Max-Planck-Institut für molekulare Zellbiologie und Genetik (MPI-CBG) haben nun ein Modell entwickelt, mit dem man Verzerrungen beseitigen und zuverlässige Aussagen treffen kann. Szabolcs Horvát erläutert: „Wir haben ein Nullmodell für verbundene Netzwerke entwickelt, bei dem der Verzerrungseffekt unter Kontrolle ist und herausgerechnet werden kann. Konkret haben wir einen Algorithmus entwickelt, der zufällig verbundene Netzwerke mit einer vorgeschriebenen Anzahl von Verbindungen für jeden Knoten generieren kann. Mit unserer Methode zeigen wir, dass die üblichen, naiven Ansätze zu ungültigen Ergebnissen führen können.“ Der Koordinator der Studie, Carl Modes, fasst zusammen: „Dieses Ergebnis verdeutlicht den Bedarf an mathematisch fundierten Methoden. Wir hoffen, dass unsere Studie für die breite Community der Netzwerkwissenschaftler nützlich sein wird. Um es anderen Forschern so einfach wie möglich zu machen, sie zu nutzen, haben wir auch eine Software entwickelt und offen zugänglich gemacht.“
Frei verfügbare Software:
https://github.com/szhorvat/ConnectedGraphSampler
Szabolcs Horvát and Carl D. Modes, Connectedness matters: Construction and exact random sampling of connected networks, J. Phys. Complex 2, 015008 (2021).