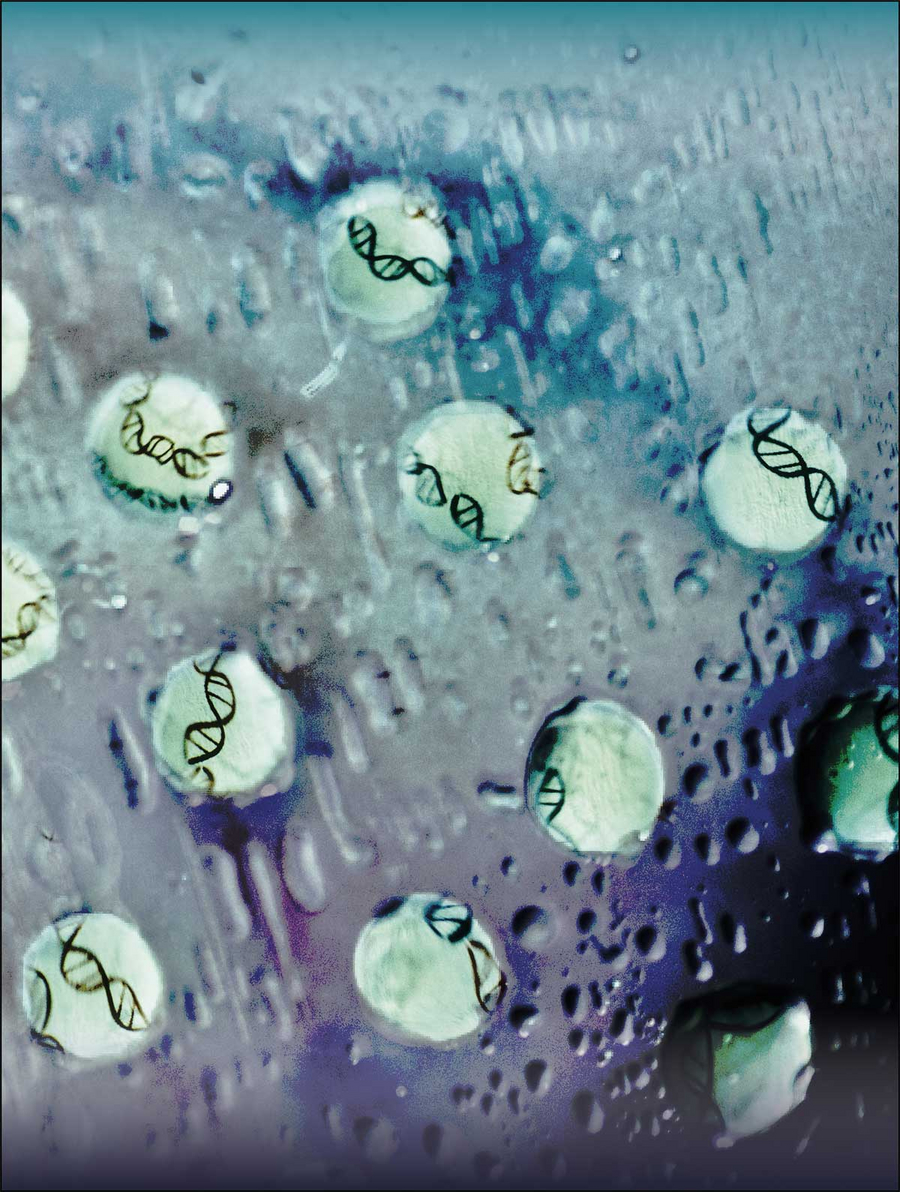
Tröpfchen aus Transkriptionsfaktoren benetzen eine Oberfläche und enthüllen regulatorische DNA-Regionen. Dieser Prozess wird durch dieses Foto symbolisiert, welches Tröpfchen von gereinigtem Protein auf einer Glasoberfläche mit handgezeichneter DNA auf der Rückseite des Glases zeigt. Copyright: Mark Leaver, Jose A. Morin and Sina Wittmann, / MPI-CBG / Jose A. Morin et al. Nature Physis, 2022
Während der Entwicklung eines Organismus teilt sich eine Zelle viele Male, um die verschiedenen Gewebe des erwachsenen Körpers zu bilden. Dabei bekommen alle Zellen das gleiche genetische Material (DNA). In einem Prozess, der als Transkription bezeichnet wird, werden Teile der DNA, die Gene, in ein Botenmolekül, die Ribonukleinsäure (RNA), kopiert. Diese wird benutzt, um Proteine herzustellen, die Bausteine unserer Körper. Welche Teile unserer DNA gelesen und abgeschrieben werden, bestimmt das Schicksal unserer Zellen. Das Lesen der DNA wird von bestimmten Proteinen, den Transkriptionsfaktoren, ausgeführt: Sie binden an bestimmte Stellen auf der DNA und aktivieren dadurch den Transkriptionsprozess. Bisher war ungeklärt, wie sie erkennen, an welche Stelle sie genau binden müssen und wie diese von anderen zufälligen Bindungsstellen im Genom unterschieden werden. Forschende des Max-Planck-Instituts für molekulare Zellbiologie und Genetik (MPI-CBG) und des Max-Planck-Instituts für Physik komplexer Systeme (MPI-PKS), beide in Dresden, zeigen, dass hierfür Tausende von Transkriptionsfaktoren als Team zusammenarbeiten und miteinander wechselwirken. Sie benetzen gemeinsam die DNA-Oberfläche, indem sie flüssige Tropfen bilden, die Ansammlungen von Bindestellen auf der DNA-Oberfläche erkennen können.
Transkription, einer der grundlegendsten zellulären Prozesse, ist der Vorgang, bei dem die in der DNA enthaltene Information in das Botenmolekül RNA transkribiert wird. Diese „Botschaft“ wird später in Proteine übersetzt. Die Entscheidung, welche Teile der DNA zu einem bestimmten Zeitpunkt transkribiert werden sollen, ist von entscheidender Bedeutung für die richtige Entwicklung eines Organismus, um dessen Gesundheit zu erhalten. Wenn die genetischen Programme nicht korrekt ausgeführt werden, steigt die Wahrscheinlichkeit, dass verschiedene Krankheiten entstehen. Die Entscheidung, welche Gene transkribiert werden, wird von einem komplexen Netzwerk regulatorischer Proteine getroffen, welche als Transkriptionsfaktoren bezeichnet werden. Während diese Faktoren lediglich an kurze DNA-Sequenzen binden, ist die Erkennung von Ansammlungen solcher Sequenzen erforderlich, um nahe gelegene Gene anzuschalten.
Die Forschungsgruppen von Stephan Grill und Anthony Hyman, beide Direktoren am MPI-CBG, und die Gruppe von Frank Jülicher, Direktor am MPI-PKS, untersuchten in ihrer aktuellen Studie in der Fachzeitschrift Nature Physics, wie Transkriptionsfaktoren die Ansammlungen vieler spezifischer DNA-Sequenzen finden und erkennen können, daran binden und auf diese Weise Gene aktivieren können. Dabei verfolgten die Forschenden einen interdisziplinären Ansatz, der experimentelle und theoretische Biophysik mit Zellbiologie verbindet. Jose Morin, einer der Erstautoren der Studie, erklärt: „Wir verwendeten optische Pinzetten eine Technologie, bei der Laser eingesetzt werden, um sehr kleine Objekte wie beispielsweise ein einzelnes DNA-Moleküle zu greifen und festzuhalten. Damit können wir dann beobachten, wie Transkriptionsfaktoren an ihre bevorzugten DNA-Sequenzen binden und Proteinkondensate bilden. Die Tatsache, dass wir diesen Prozess an einzelnen isolierten Molekülen untersuchen können, ermöglicht es uns, einzelne Interaktionen sichtbar zu machen die sich ansonsten innerhalb einer Zelle, aufgrund Millionen andere Proteine die dort präsent sind, verlieren würden.“ Sina Wittmann, eine weitere Erstautorin, ergänzt: „Die Physiker in unserem Team haben uns geholfen zu verstehen, wie Transkriptionsfaktoren miteinander kommunizieren und sich in Teamarbeit zusammenfinden. Sie durchlaufen einen Übergang zu einer sogenannten Vorbenetzung, bei dem sie flüssigkeitsähnliche Tröpfchen bilden, vergleichbar mit den Tropfen die auf dem Badezimmerspiegel beim Duschen entstehen. Diese Kondensate sind mit Tausenden von Transkriptionsfaktoren gefüllt. Auf diese Weise zusammengefügt, können die Transkriptionsfaktoren nun die richtige DNA-Region identifizieren, indem sie gemeinsam die DNA-Sequenz auslesen.“
Stephan Grill fasst zusammen: „Wir haben jetzt eine mögliche mechanistische Erklärung für die Lokalisierung von Transkriptionsfaktoren entlang des Genoms. Dies ist wichtig, um zu verstehen, wie die Genexpression reguliert wird. Da wir wissen, dass diese Regulation bei Entwicklungskrankheiten und Krebs gestört ist, geben uns diese neuen Ergebnisse ein deutlicheres Bild davon, wie diese Krankheiten entstehen. Dieses Wissen ist wichtig, um über neue therapeutische Ansätze nachzudenken, welche die Teamarbeit der Transkriptionsfaktoren berücksichtigen.“
Die Forschungsgruppen von Anthony Hyman, Stephan Grill und Frank Jülicher sind außerdem dem Zentrum für Systembiologie Dresden (CSBD) und dem Exzellenzcluster "Physics of Life" (PoL) der TU Dresden angegliedert. Das CSBD ist eine Kooperation zwischen dem MPI-CBG, dem MPI-PKS und der TU Dresden. In dem interdisziplinären Zentrum arbeiten Physiker, Informatiker, Mathematiker und Biologen zusammen, um zu verstehen, wie Zellen ihr Verhalten koordinieren, um Gewebe und Organe einer bestimmten Form oder Funktion zu bilden. Das Exzellenzcluster PoL der TU Dresden erforscht die physikalischen Gesetze, die der Selbstorganisation des Lebens in Molekülen, Zellen und Geweben zugrunde liegen. In dem von der Deutschen Forschungsgemeinschaft (DFG) geförderten Cluster untersucht ein interdisziplinäres Team von Wissenschaftlern gemeinsam, wie sich aktive lebende Materie selbst organisiert, um Leben entstehen zu lassen.
Jose A. Morin, Sina Wittmann, Sandeep Choubey, Adam Klosin, Stefan Golfier, Anthony A. Hyman, Frank Jülicher, Stephan W. Grill: Sequence dependent surface condensation of a pioneer transcription factor on DNA. Nature Physics. Doi: 10.1038/s41567-021-01462-2
Pressekontakt MPI-CBG
Katrin Boes
+49 (0) 351 210 2080
kboes(at)mpi-cbg.de
Weitere Informationen:
Prof. Stephan Grill
grill(at)mpi-cbg.de