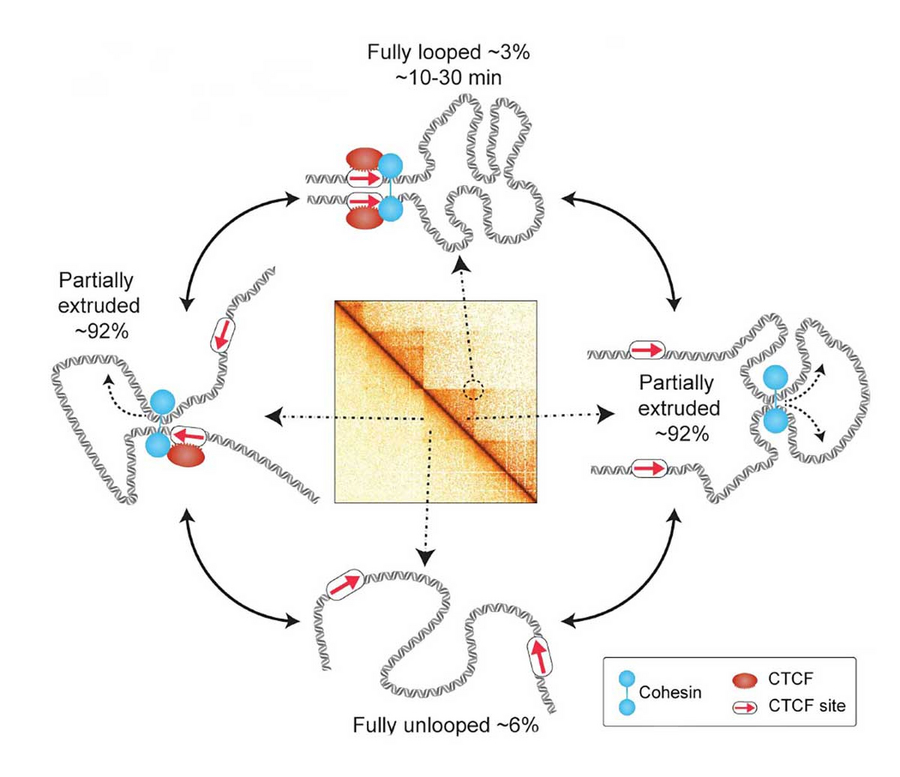
Die superauflösende Live-Zell-Bildgebung von CTCF/Cohesin-vermittelten Chromatinschlaufen zeigt, dass diese Schlaufen sowohl selten als auch dynamisch sind. © Gabriele et al., Science 10.1126/science.abn6583 (2022).
In menschlichen Chromosomen ist die DNA von Proteinen umhüllt, sodass eine sehr lange Faser entsteht. Diese Faser ist in zahlreiche Schlaufen zusammengefaltet, von denen man vermutet, dass sie unter anderem die DNA-Reparatur erleichtern und den Zellen helfen, ihre Genexpression zu steuern. Eine neue Studie des Massachusetts Institute of Technology (MIT), des Max-Planck-Instituts für molekulare Zellbiologie und Genetik (MPI-CBG) und des Zentrums für Systembiologie Dresden (CSBD) deutet darauf hin, dass diese Schlaufen sehr dynamisch sind und kurzlebiger als bisher angenommen. In der neuen Studie konnten Forschende die Bewegung eines Abschnitts des Genoms einer lebenden Zelle über einen Zeitraum von etwa zwei Stunden beobachten. Sie stellten fest, dass dieser Abschnitt nur für etwa drei bis sechs Prozent der Zeit vollständig zu einer Schlaufe geformt war und nur etwa 10 bis 30 Minuten in diesem Zustand verblieb. Die neuen Ergebnisse deuten darauf hin, dass das gegenwärtige Verständnis davon, wie Schleifen die Genexpression beeinflussen, möglicherweise überarbeitet werden muss, sagen die Forschenden.
„Viele Modelle in diesem Bereich zeigen ein Bild von statischen Schlaufen, die diese Prozesse regulieren. Unsere neue Studie zeigt, dass dieses Bild nicht wirklich korrekt ist“, sagt Anders Sejr Hansen, Underwood-Prescott Career Development Assistant Professor for Biological Engineering am MIT. „Wir vermuten, dass der funktionsrelevante Zustand dieser Domänen sehr viel dynamischer ist.“ Hansen ist einer der Seniorautoren der neuen Studie, zusammen mit Leonid Mirny, Professor am Institute for Medical Engineering and Science des MIT und des Departments für Physik und Christoph Zechner, Gruppenleiter am MPI-CBG und am CSBD. Die Erstautoren der Studie, die heute in Science erscheint, sind Michele Gabriele,Post-Doktorand am MIT, Hugo Brandão, der kürzlich von der Harvard University promoviert wurde und MIT Doktorand Simon Grosse-Holz.
Außerhalb der Schlaufe
Anhand von Computersimulationen und experimentellen Daten haben Wissenschaftlerinnen und Wissenschaftler, darunter auch die Gruppe von Leonid Mirny am MIT, gezeigt, dass Schlaufen im Genom durch einen Prozess entstehen, der Extrusion genannt wird und bei dem ein molekularer Motor für das kontinuierliche Wachstum dieser Schlaufen verantwortlich ist. Der Motor bleibt stehen, wenn er auf ein „Stopp-Schild“ auf der DNA trifft. Dieser Motor ist ein Proteinkomplex namens Cohesin, während das DNA-gebundene Protein CTCF als Stopp-Schild dient. Die Cohesin-abhängigen Schlaufen zwischen CTCF-Bindestellen wurden in früheren Experimenten beobachtet. Allerdings lieferten diese bisherigen Experimente nur eine Momentaufnahme, ohne Informationen darüber, wie sich die Schlaufen im Laufe der Zeit verändern. In ihrer neuen Studie entwickelten die Forschenden Techniken, mit denen sie die CTCF-Bindestellen auf der DNA fluoreszent markieren konnten, sodass sie die DNA-Schlaufen über mehrere Stunden hinweg abbilden konnten. Außerdem entwickelten sie eine neue Analysemethode, mit der sie aus den Bilddaten auf den Zustand der Schlaufe schließen konnten. “Diese Methode war für uns entscheidend, um Signal und Rauschen in unseren experimentellen Daten zu unterscheiden und die Schlaufenbildung zu quantifizieren“, sagt Christoph Zechner. „Wir glauben, dass solche Ansätze für die Biologie zunehmend wichtiger werden, da wir experimentell immer näher an die Detektionsgrenze gehen.“
Die Forschenden nutzten ihre Methode, um einen Abschnitt des Genoms in embryonalen Stammzellen der Maus bildlich darzustellen. Simon Grosse-Holz erklärt: „Im Kontext eines gesamten Zellteilungszyklus, der etwa 12 Stunden dauert, existieren die vollständig ausgebildeten Schlaufen nur etwa 3 bis 6 Prozent der Zeit, also etwa 20 bis 45 Minuten.“
„Wenn die Schlaufe nur für einen so kleinen Zeitraum des Zellzyklus vorhanden ist, können wir nicht davon ausgehen, dass dieser vollständig ausgebildete Schlaufenzustand der primäre Regulator der Genexpression ist“, sagt Hansen. „Wir glauben, dass wir neue Modelle dafür brauchen, wie die 3D-Struktur des Genoms die Genexpression, die DNA-Reparatur und andere davon abhängige Prozesse reguliert.“ Während vollständig ausgebildete Schlaufen selten waren, fand das Forscherteam heraus, dass in etwa 92 Prozent der Fälle teilweise etablierte Schlaufen vorhanden waren. Diese kleineren Schlaufen waren mit den bisherigen Methoden zum Nachweis von Schlaufen im Genom nur schwer zu beobachten. „In dieser Studie konnten wir durch die Integration unserer experimentellen Daten mit Polymersimulationen nun die relativen Anteile der vollständig zur Schlaufe geformten, teilweise gefalteten, und vollkommen offenen Zustände quantifizieren“, ergänzt Hugo Brandão.
„Da diese Interaktionen sehr kurz, aber sehr häufig sind, konnten die bisherigen Methoden ihre Dynamik nicht vollständig erfassen“, sagt Gabriele. „Mit unserer neuen Technik können wir die Übergänge zwischen vollständig ausgebildeten Schlaufen und offenen Konformationen auflösen.“
Die Autoren der Studie vermuten, dass diese unvollständigen Schlaufen bei der Genregulierung eine wichtigere Rolle spielen könnten als die vollständig ausgebildeten. Wenn sich Schlaufen bilden und wieder lösen, laufen DNA-Stränge aneinander vorbei; diese Interaktionen können regulatorischen Elementen wie Enhancern und Genpromotoren helfen, zueinander zu finden. „Mehr als 90 Prozent der Zeit gibt es transiente Schlaufen, und vermutlich ist es entscheidend, dass es diese Schlaufen gibt, die ständig neu ausgebildet werden“, sagt Mirny. „Der Prozess der Extrusion selbst könnte wichtiger sein als der Zustand der vollständigen Schlaufe, den es nur für eine sehr kurze Zeit gibt.“
Untersuchung von weiteren Schlaufen
Da die meisten anderen Schlaufen im Genom schwächer sind als die von den Forschenden untersuchte, vermuten sie, dass diese anderen Schlaufen sich ebenfalls als sehr kurzlebig erweisen werden. Das Forscherteam plant nun, mit ihrer neuen Technik einige dieser anderen Schlaufen in verschiedenen Zelltypen zu untersuchen. „Es gibt etwa 10.000 dieser Schlaufen, und wir haben uns eine davon angesehen“, sagt Hansen. „Wir haben viele indirekte Hinweise darauf, dass die Ergebnisse verallgemeinerbar sind, aber wir haben das noch nicht zeigen können. Mit der von uns entwickelten Technologieplattform, die neue experimentelle Methoden und Rechenverfahren kombiniert, können wir uns nun andere Schlaufen im Genom ansehen.“ Des weiteren planen sie die Rolle bestimmter Schlaufen bei Krankheiten zu untersuchen. Viele Krankheiten, darunter eine neurologische Entwicklungsstörung namens FOXG1-Syndrom, könnten mit einer fehlerhaften Schlaufendynamik zusammenhängen. Die Forschenden untersuchen gerade, wie sowohl die normale als auch die mutierte Form des FOXG1-Gens sowie das krebsverursachende Gen MYC von der Schlaufenbildung im Genom beeinflusst werden.
Die Forschung wurde von den National Institutes of Health, der National Science Foundation, der Mathers Foundation, einem Pew-Stewart Cancer Research Scholar Stipendium, den Chaires d'excellence Internationale Blaise Pascal, einem Forschungsstipendium der American-Italian Cancer Foundation und dem Max-Planck-Institut für molekulare Zellbiologie und Genetik finanziert.
Video zur Darstellung der Chromosomenorganisation durch Schlaufenextrusion mit Barriereelementen aus dem Mirny-Labor: https://www.youtube.com/watch?v=8FW6gOx5lPI
Mit Genehmigung von MIT News veröffentlicht.
Michele Gabriele, Hugo B. Brandão, Simon Grosse-Holz, Asmita Jha, Gina M. Dailey, Claudia Cattoglio, Tsung-Han S. Hsieh, Leonid Mirny, Christoph Zechner, Anders S. Hansen: Dynamics of CTCF and cohesin mediated chromatin looping revealed by live-cell imaging. Science, 14. April 2022, doi: 10.1126/science.abn6583
Anders Sejr Hansen
+1 617-253-6086
ashansen(at)mit.edu
Christoph Zechner
+49 (0) 351 210-2735
zechner(at)mpi-cbg.de