Biologen sind nie wirklich zufrieden mit den Bildern, die ihre Mikroskope liefern. Sie könnten schärfer sein, mehr Detail zeigen und weniger Bildrauschen aufweisen. Neben der Möglichkeit die Mikroskope selbst zu verbessern, können Computer helfen, die Unschärfe in Bildern zu korrigieren. Eine computergestützte Technik, die versucht das real zugrundeliegende Bild zu rekonstruieren, ist die Bildentfaltung (engl. Deconvolution). Die Ergebnisse können beeindruckend sein, allerdings ist die Berechnung sehr aufwändig, insbesondere für große dreidimensionale Bilder, die über einen langen Zeitraum hinweg aufgenommen werden.
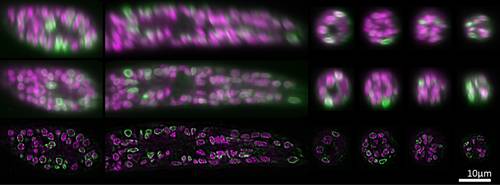
Stephan Preibisch aus der Forschungsgruppe von Pavel Tomancak (MPI-CBG), der momentan mit Eugene Myers und Robert Singer arbeitet, wollte sich mit den existierenden Lösungen nicht abfinden und die Technik verbessern. Dazu verwendete er Daten von Mikroskopen, die eine Betrachtung von biologischen Proben aus verschiedenen Winkeln erlaubt. Diese so genannte multiview Mikroskopie macht das komplexe Problem des Abschätzens des zugrundeliegenden realen Bildes besser lenkbar. Preibisch fand heraus, dass es möglich ist, den Berechnungsprozess um ein Vielfaches zu beschleunigen, indem man die Abhängigkeiten zwischen den verschiedenen Bildern derselben Probe in die Methode integriert. Gemeinsam mit Fernando Amat vom HHMI Janelia Farm Campus entwickelte er einen Algorithmus und eine Software welche zusätzlich die Rechenleistung von modernen Grafikkarten nutzt und damit erlaubt die multiview Entfaltung innerhalb von Minuten statt Stunden zu berechnen. Die Ergebnisse sind überwältigend. Auf einer multiview Abbildung von Larven des Fadenwurms C. elegans kann man mühelos die Zellkerne voneinander unterscheiden. Diese Algorithmen ließen sich auch bei anderen Modelorganismen anwenden, wie z.B. bei stundenlangen Aufnahmen der Embryonalentwicklung von Drosophila mit einem SPIM Mikroskop (Selective Plane Illumination Microscope). Preibischs Ansatz ist der einzige, der in der Lage ist, so große Datensätze in akzeptabler Zeit zu bearbeiten.
Seine Algorithmen können bei verschiedensten Mikroskopen angewendet werden, da es sich um eine Open Source Software handelt. Die Software ist als Plug-In für die Fiji Plattform erhältlich. Die Ergebnisse wurden in der Fachzeitschrift Nature Methods veröffentlicht.